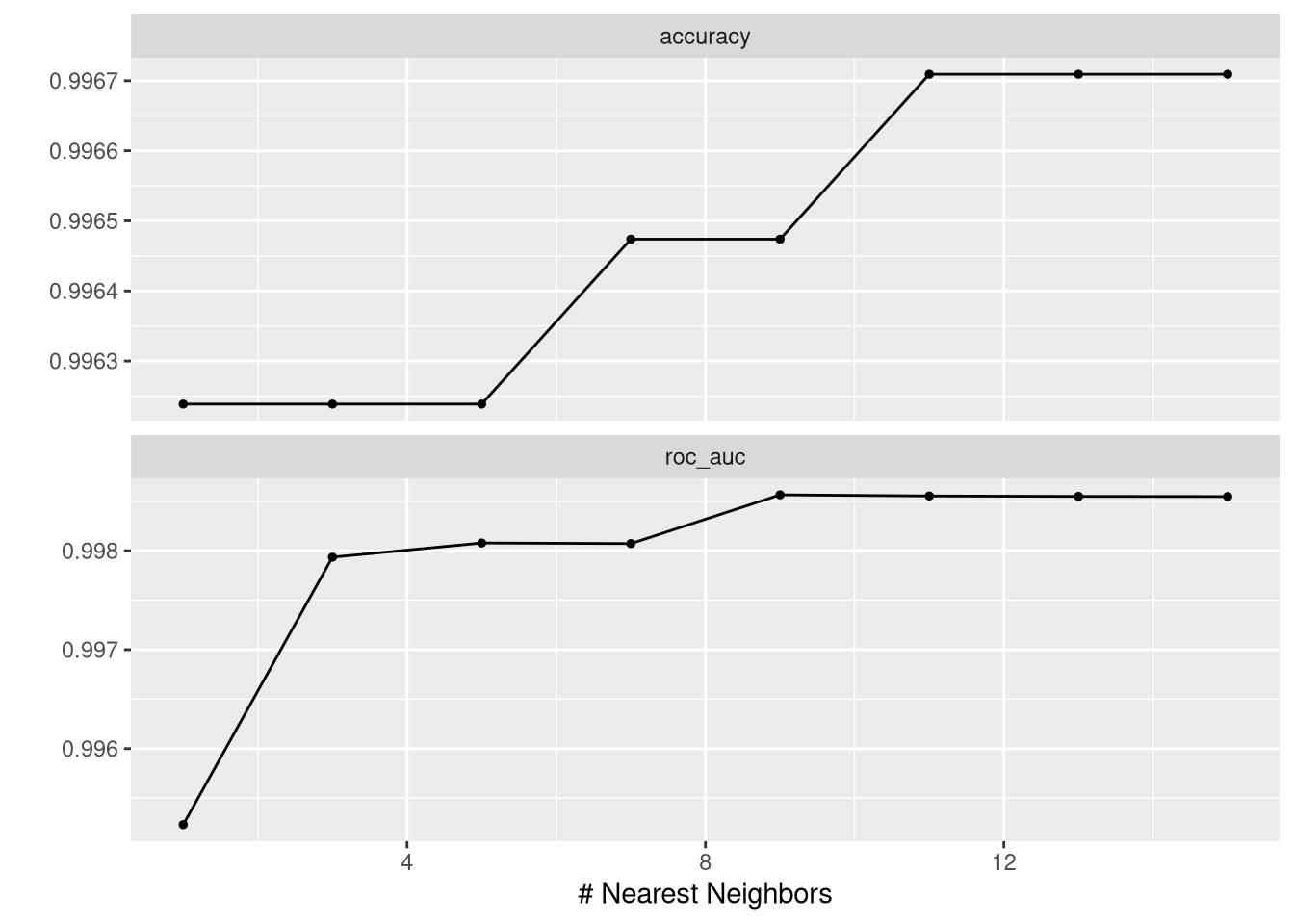
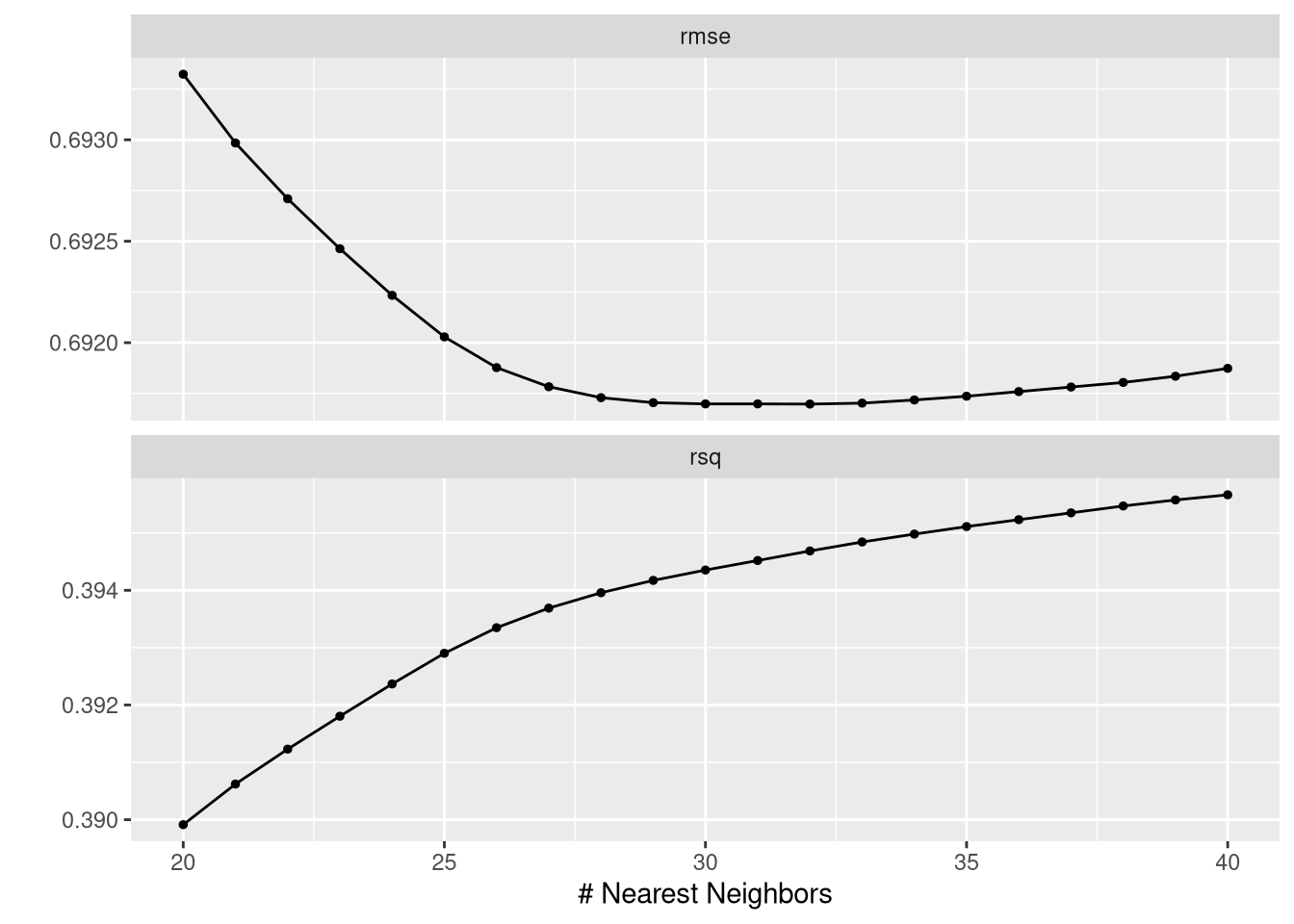
Rows: 5,320
Columns: 14
$ tipo <fct> blanco, blanco, blanco, blanco, blanco, blanco, b…
$ meses_barrica <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
$ acided_fija <dbl> 7.0, 6.3, 8.1, 7.2, 6.2, 8.1, 8.1, 8.6, 7.9, 6.6,…
$ acided_volatil <dbl> 0.27, 0.30, 0.28, 0.23, 0.32, 0.22, 0.27, 0.23, 0…
$ acido_citrico <dbl> 0.36, 0.34, 0.40, 0.32, 0.16, 0.43, 0.41, 0.40, 0…
$ azucar_residual <dbl> 20.70, 1.60, 6.90, 8.50, 7.00, 1.50, 1.45, 4.20, …
$ cloruro_sodico <dbl> 0.045, 0.049, 0.050, 0.058, 0.045, 0.044, 0.033, …
$ dioxido_azufre_libre <dbl> 45, 14, 30, 47, 30, 28, 11, 17, 16, 48, 41, 28, 3…
$ dioxido_azufre_total <dbl> 170, 132, 97, 186, 136, 129, 63, 109, 75, 143, 17…
$ densidad <dbl> 1.0010, 0.9940, 0.9951, 0.9956, 0.9949, 0.9938, 0…
$ ph <dbl> 3.00, 3.30, 3.26, 3.19, 3.18, 3.22, 2.99, 3.14, 3…
$ sulfatos <dbl> 0.45, 0.49, 0.44, 0.40, 0.47, 0.45, 0.56, 0.53, 0…
$ alcohol <dbl> 8.8, 9.5, 10.1, 9.9, 9.6, 11.0, 12.0, 9.7, 10.8, …
$ calidad <int> 6, 6, 6, 6, 6, 6, 5, 5, 5, 7, 5, 7, 6, 8, 6, 5, 7…