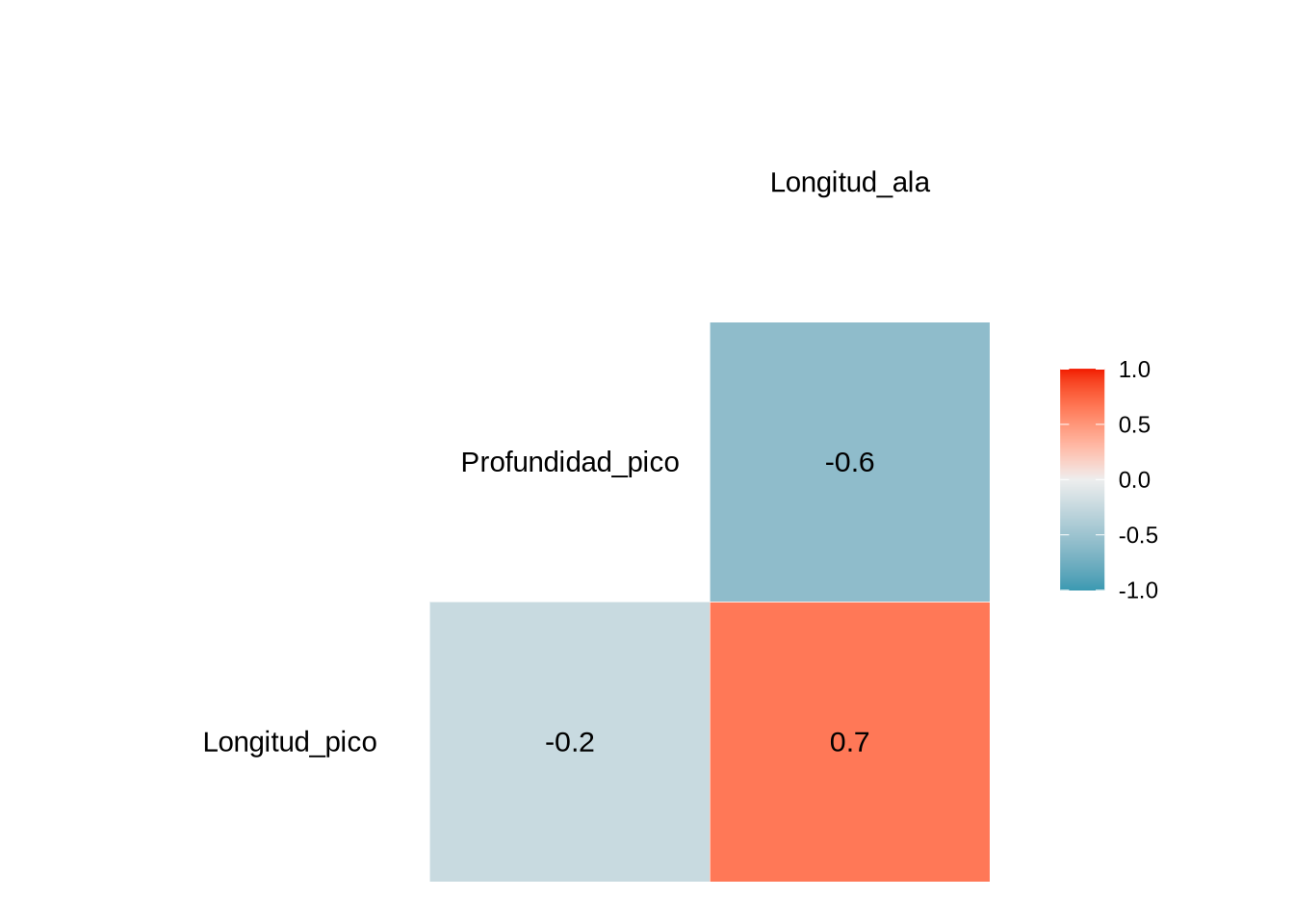
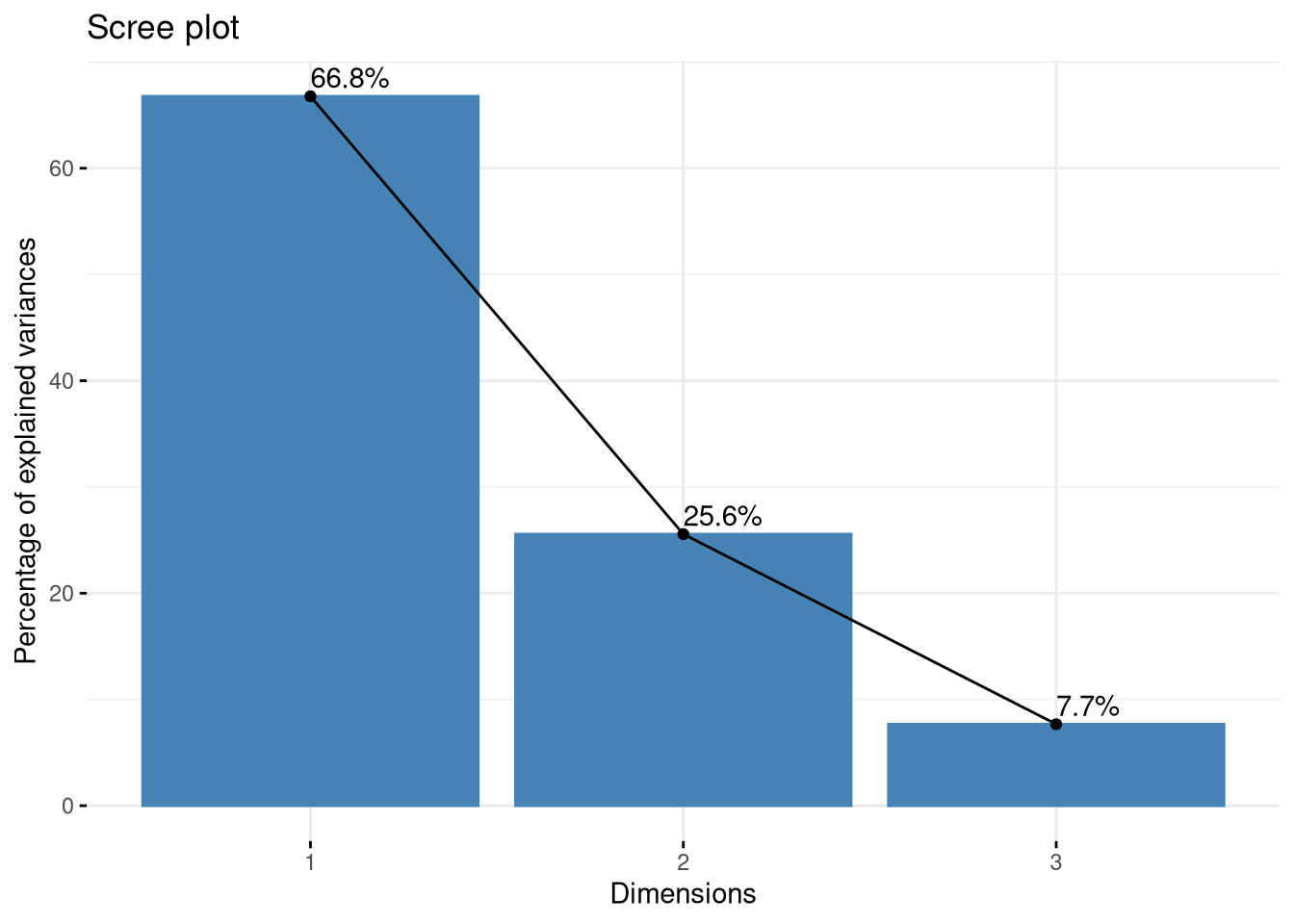
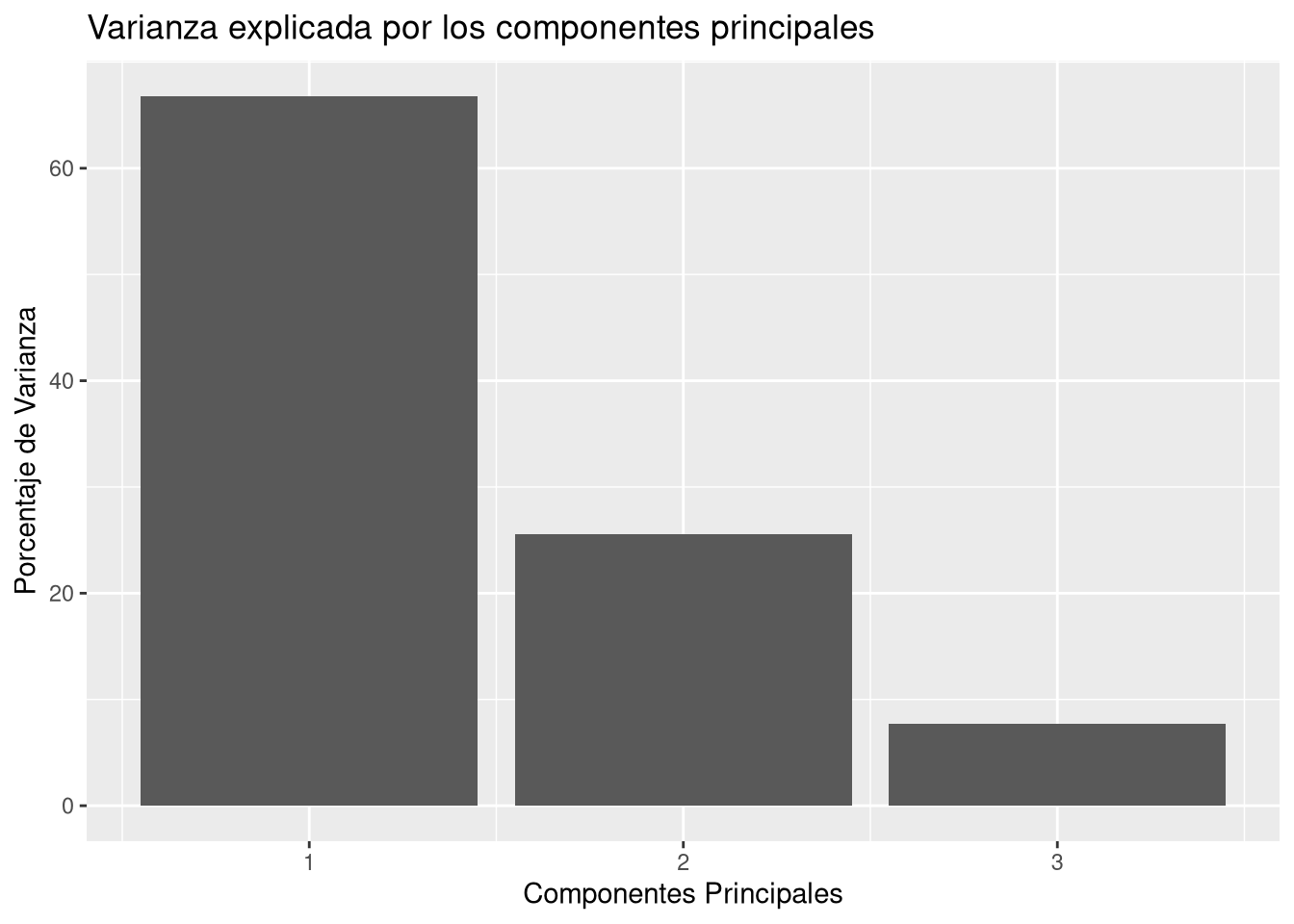
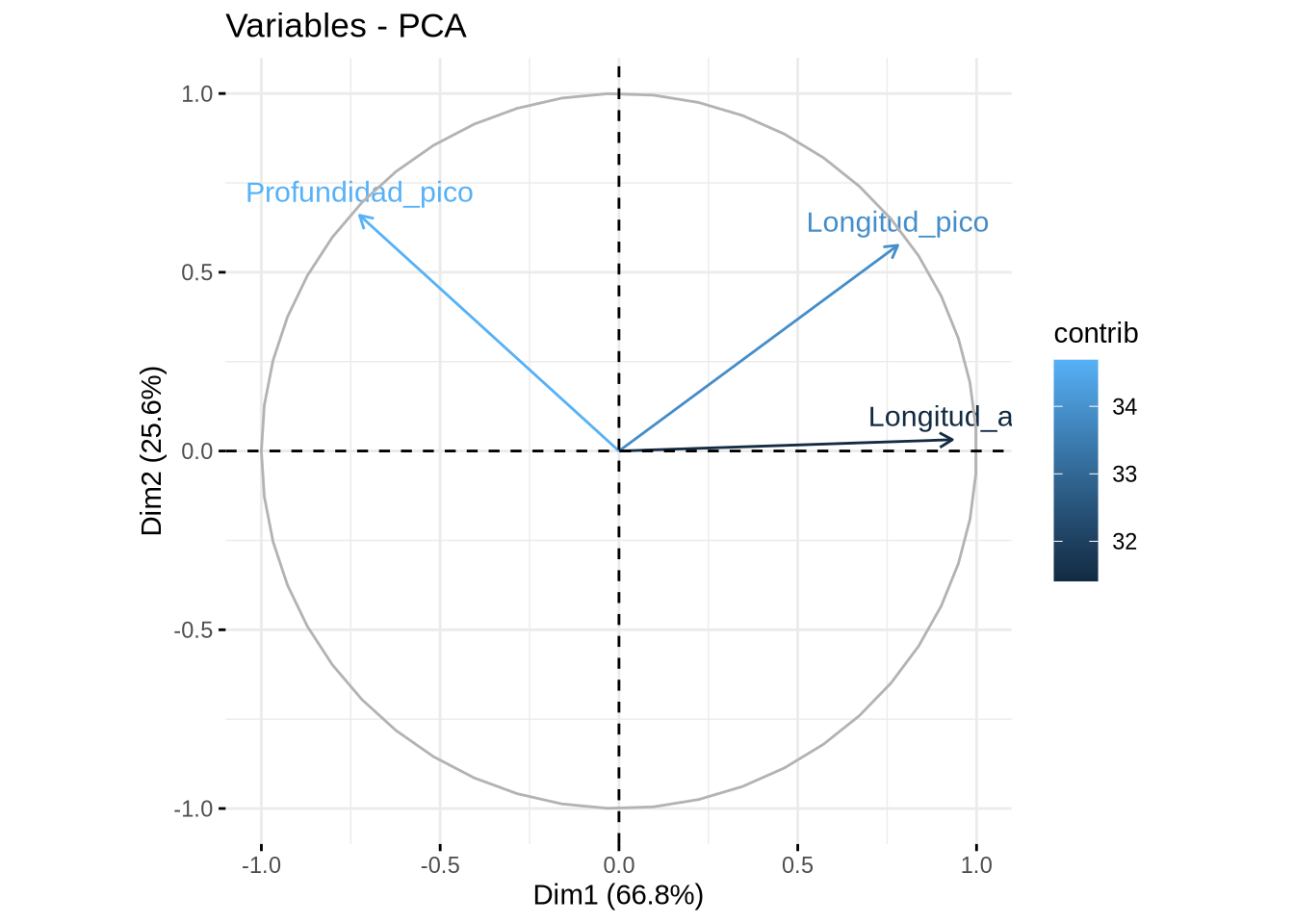
Rows: 991
Columns: 31
$ Id <fct> 28-2017-11-28-L, 90-2017-11-24-L, 106-2017-10-25-L, 170-2…
$ Ojo <fct> I, I, I, I, I, I, I, I, I, I, I, I, I, I, I, I, I, I, I, …
$ Glaucoma <fct> No, No, No, No, No, No, No, No, No, No, No, No, No, No, N…
$ AnilloBMO.G <dbl> -0.80439347, 0.19282278, -0.14995594, -0.24608299, 0.6777…
$ AnilloBMO.NS <dbl> -0.79143813, 0.36651404, 0.39448008, 0.57304030, 1.618783…
$ AnilloBMO.N <dbl> -0.52772777, -0.02541891, -0.71402835, -0.62100162, 1.025…
$ AnilloBMO.NI <dbl> -0.35277668, 0.19875609, -0.15638019, -0.89208610, -0.473…
$ AnilloBMO.TI <dbl> -0.7528896, 0.4451767, 0.6652916, -0.6153245, -0.2522384,…
$ AnilloBMO.T <dbl> -0.7439764, -0.1896643, -0.1330641, 0.3428515, 0.2142339,…
$ AnilloBMO.TS <dbl> -1.39159694, 0.75238535, 0.16532422, 0.34138752, 0.816866…
$ Anillo3.5.G <dbl> -0.09556442, 1.00585070, 0.70929372, 0.76493558, -1.40076…
$ Anillo3.5.NS <dbl> 0.33379621, 0.09117589, 0.06266397, 0.72091790, 0.4400156…
$ Anillo3.5.N <dbl> 1.28748152, 0.98044192, 0.13244712, 0.30738152, -1.045105…
$ Anillo3.5.NI <dbl> 1.15894330, -0.31496251, 0.39015716, 0.31730079, -0.94341…
$ Anillo3.5.TI <dbl> -0.50311018, 1.31127118, 1.27014639, 1.76714083, -1.79928…
$ Anillo3.5.T <dbl> -1.59142163, 0.12744265, 0.24522082, -0.79909388, -0.5739…
$ Anillo3.5.TS <dbl> -2.0908403, 1.2527074, 0.5944370, 0.8777833, -0.6874193, …
$ Anillo4.1.G <dbl> -0.121436081, 1.254674054, 0.430187973, 0.417280946, -1.2…
$ Anillo4.1.NS <dbl> 0.8613243, 0.4950556, 0.1578275, 0.5188949, 0.2699992, 0.…
$ Anillo4.1.N <dbl> 1.001114455, 1.260064455, 0.157680792, -0.156110297, -1.0…
$ Anillo4.1.NI <dbl> 1.3984707, -0.4752512, 0.1857529, 0.2147013, -0.9788313, …
$ Anillo4.1.TI <dbl> -0.2534641, 1.3862449, 1.1514398, 1.6031777, -2.3624990, …
$ Anillo4.1.T <dbl> -1.65337057, 0.37802261, 0.19480648, -1.09897557, -0.6063…
$ Anillo4.1.TS <dbl> -2.32056169, 1.18380488, 0.09630482, 1.17049000, 0.121032…
$ Anillo4.7.G <dbl> 0.08050894, 0.71157788, 0.30403470, 0.47251652, -1.301118…
$ Anillo4.7.NS <dbl> 0.82503033, 0.49344645, 0.52241046, 0.48707283, 0.0209695…
$ Anillo4.7.N <dbl> 1.15564635, 1.07853988, 0.05617859, -0.03282918, -1.07153…
$ Anillo4.7.NI <dbl> 1.25933229, -0.44207257, 0.05404354, 0.10509507, -0.59378…
$ Anillo4.7.TI <dbl> -0.07477907, 0.19373664, 1.02114279, 1.52007321, -2.53674…
$ Anillo4.7.T <dbl> -1.43681500, 0.29942282, 0.20263910, -1.08148756, -0.6537…
$ Anillo4.7.TS <dbl> -1.683414795, 0.668934521, -0.478766027, 0.886482740, 0.2…