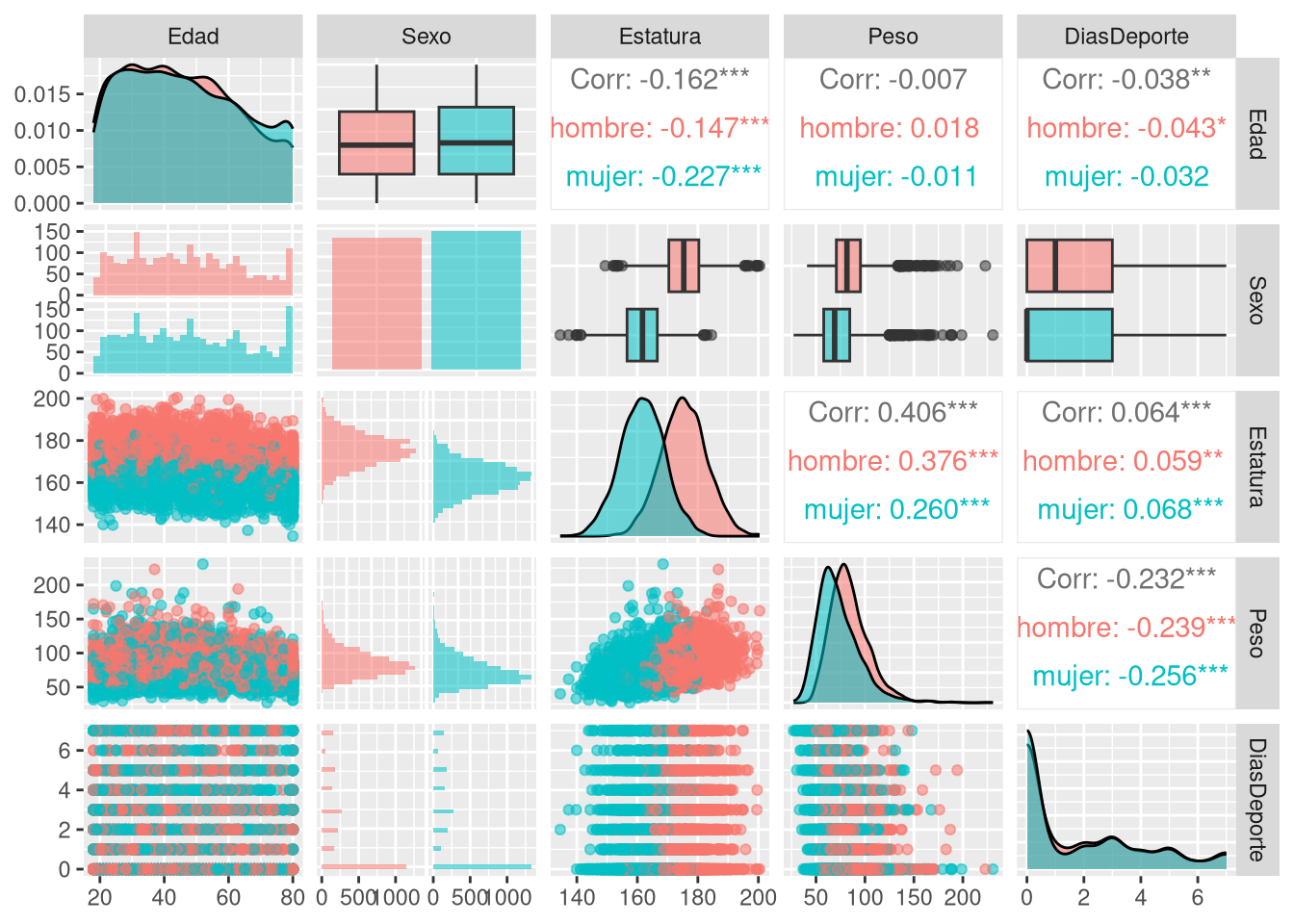
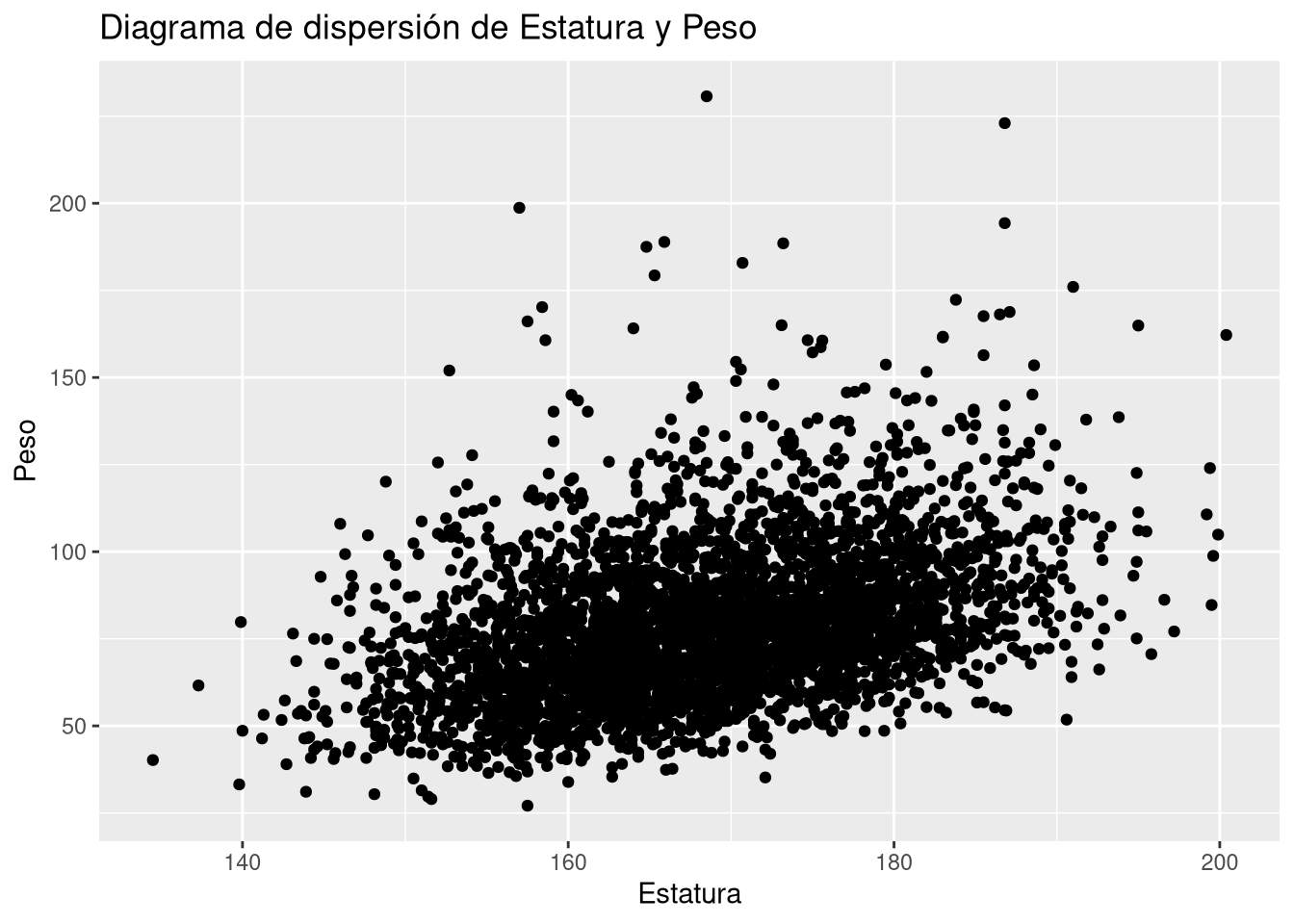
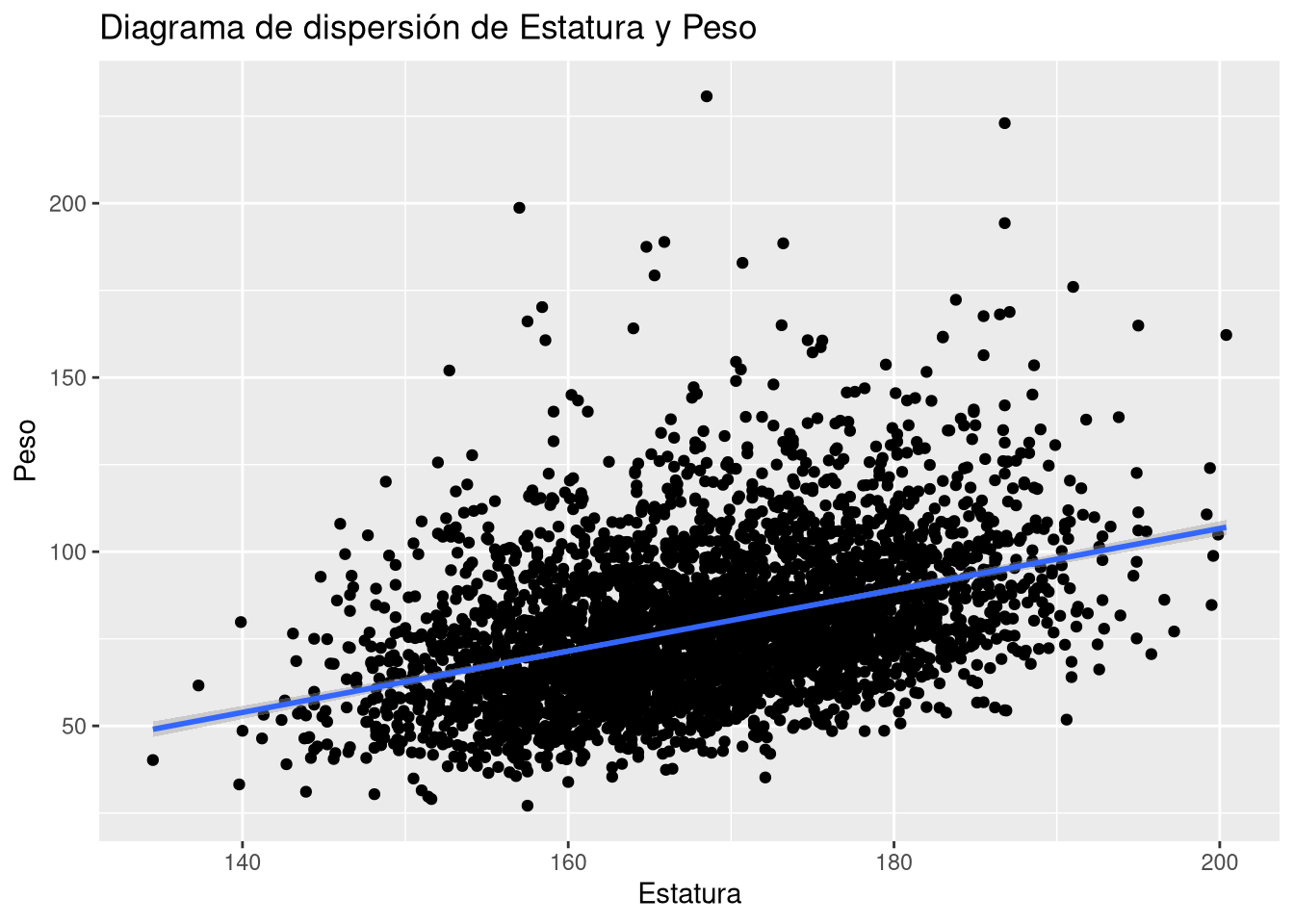
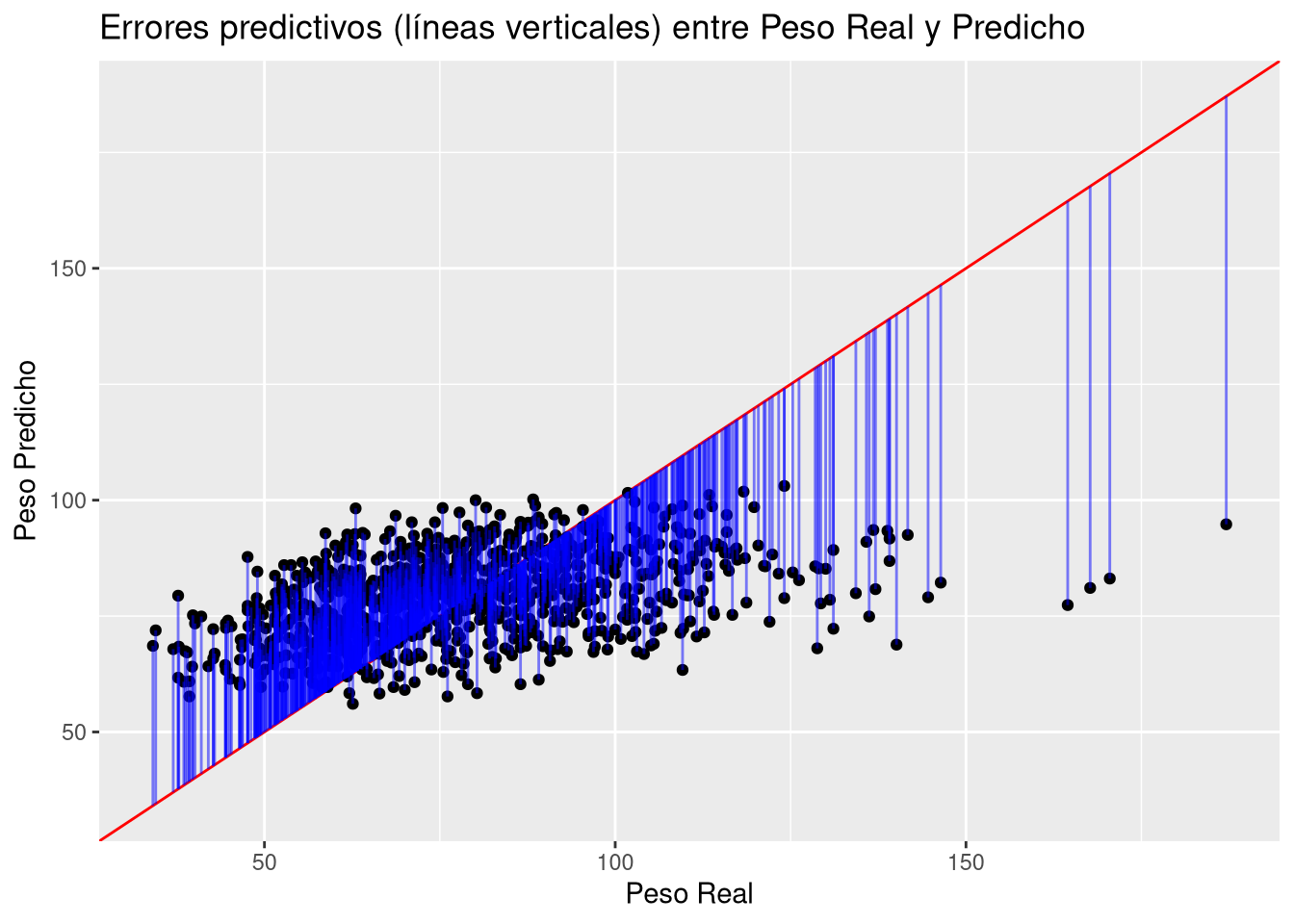
Rows: 918
Columns: 12
$ Edad <int> 40, 49, 37, 48, 54, 39, 45, 54, 37, 48, 37, 58, 39, 49…
$ Sexo <fct> H, M, H, M, H, H, M, H, H, M, M, H, H, H, M, M, H, M, …
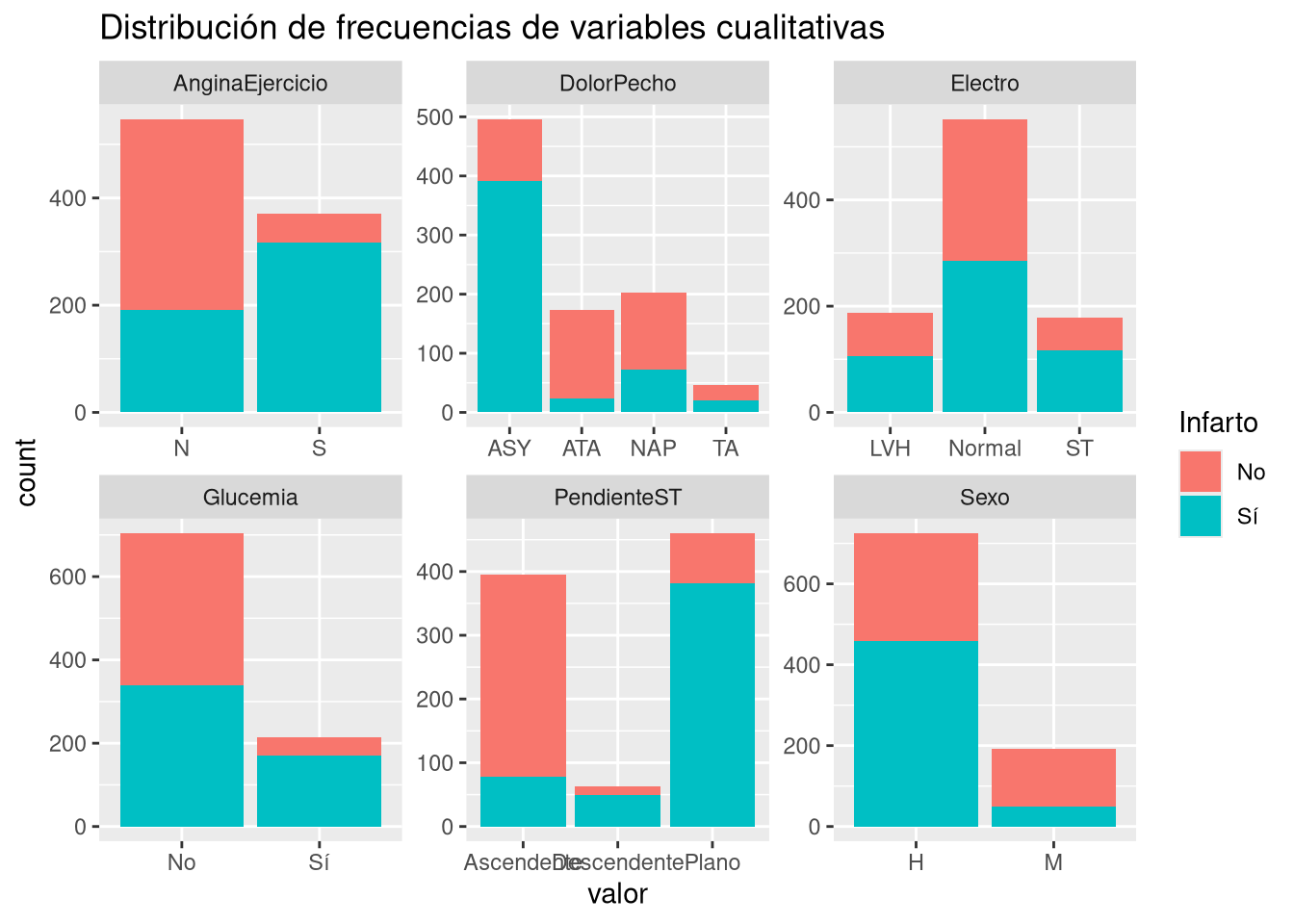
$ DolorPecho <fct> ATA, NAP, ATA, ASY, NAP, NAP, ATA, ATA, ASY, ATA, NAP,…
$ PresionArterial <int> 140, 160, 130, 138, 150, 120, 130, 110, 140, 120, 130,…
$ Colesterol <int> 289, 180, 283, 214, 195, 339, 237, 208, 207, 284, 211,…
$ Glucemia <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
$ Electro <fct> Normal, Normal, ST, Normal, Normal, Normal, Normal, No…
$ Pulsaciones <int> 172, 156, 98, 108, 122, 170, 170, 142, 130, 120, 142, …
$ AnginaEjercicio <fct> N, N, N, S, N, N, N, N, S, N, N, S, N, S, N, N, N, N, …
$ DepresionST <dbl> 0.0, 1.0, 0.0, 1.5, 0.0, 0.0, 0.0, 0.0, 1.5, 0.0, 0.0,…
$ PendienteST <fct> Ascendente, Plano, Ascendente, Plano, Ascendente, Asce…
$ Infarto <int> 0, 1, 0, 1, 0, 0, 0, 0, 1, 0, 0, 1, 0, 1, 0, 0, 1, 0, …